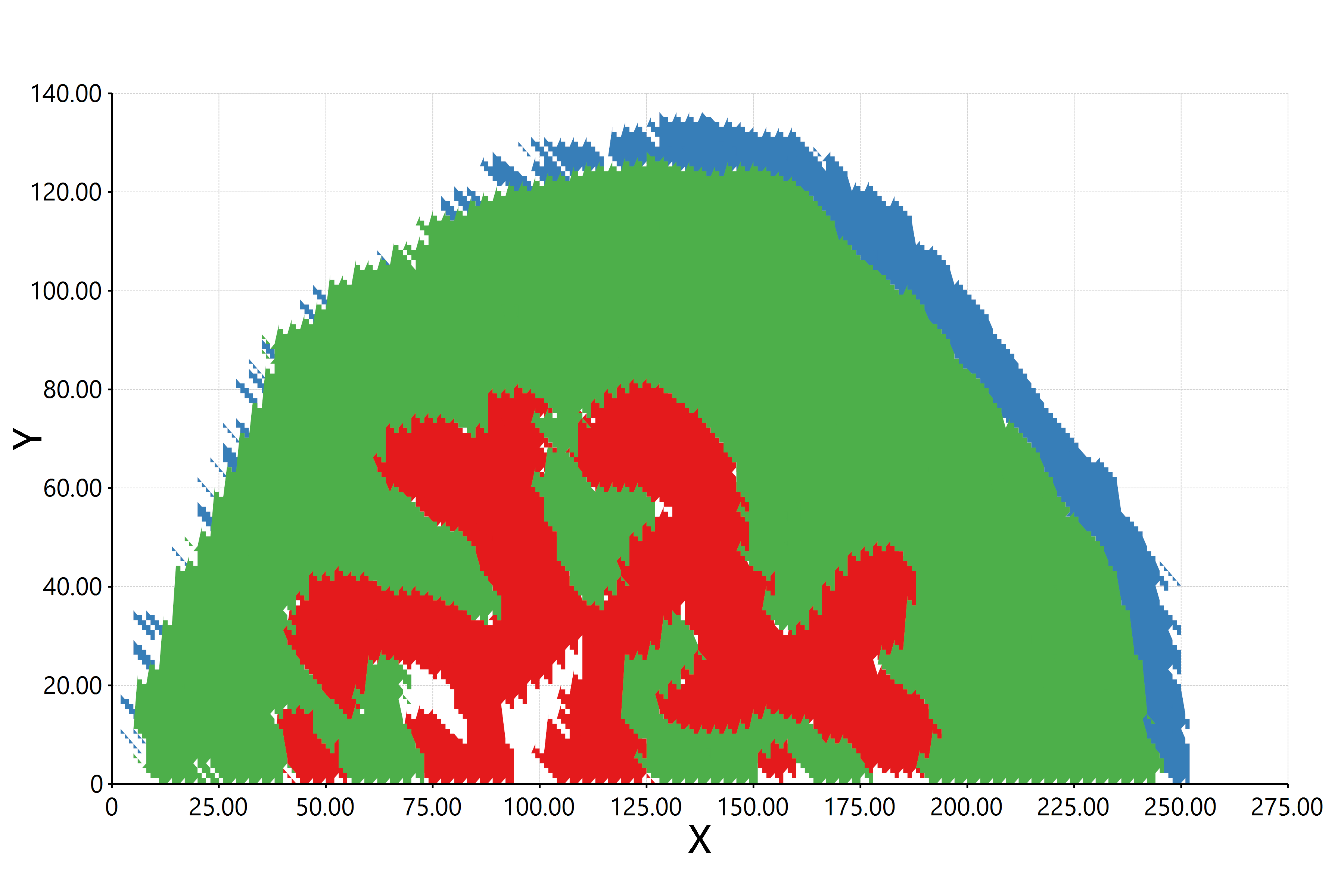
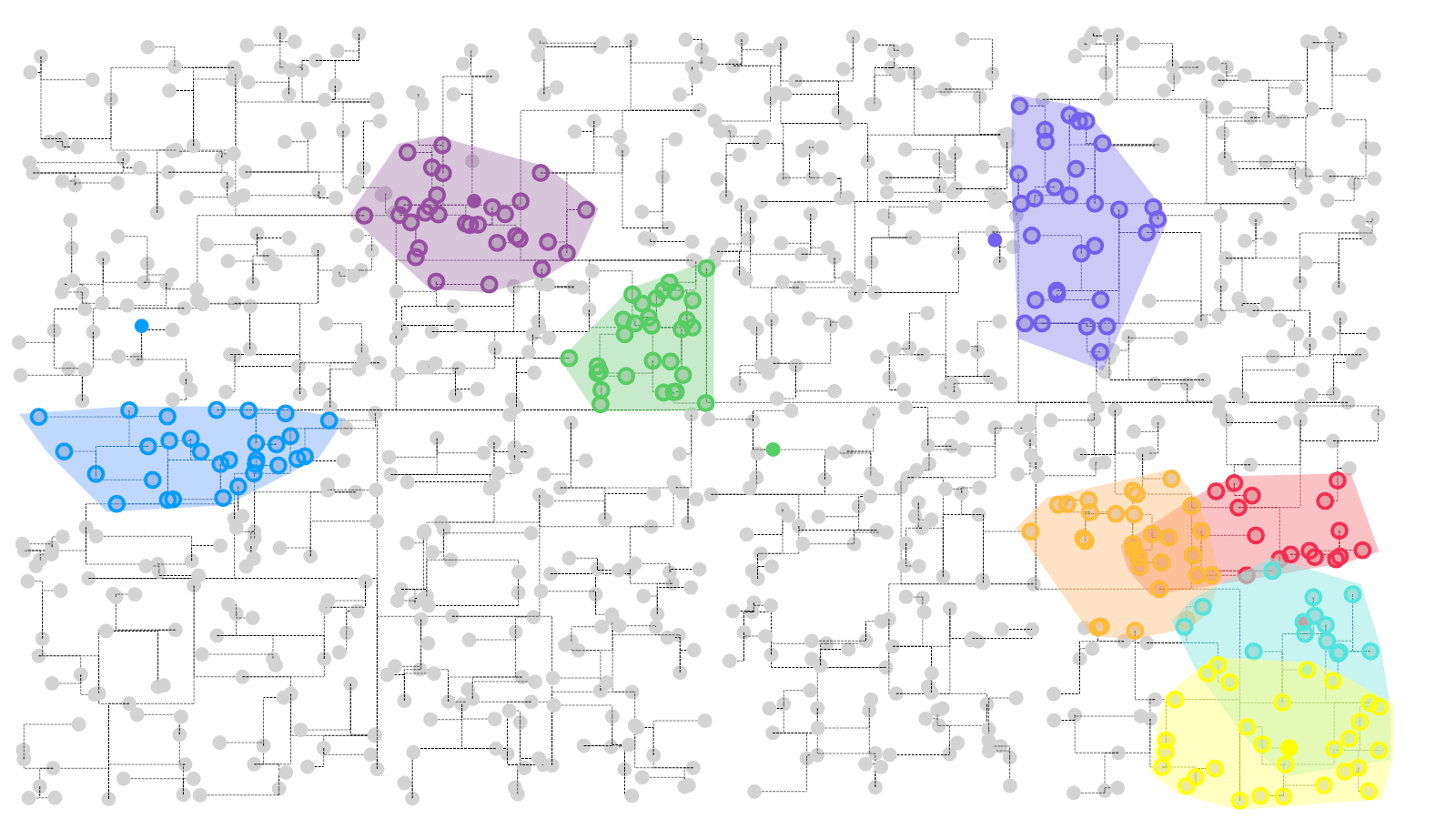
估计阅读时长: 11 分钟https://github.com/xieguigang/sciBASIC 最近在研究实现空间代谢组学中的一些特征区域的自动化划分分割。在得到了特征点集合之后,我们需要根据一些图像处理算法进行特征区域的提取操作。之前,我们尝试过基于绘制等高线图Marching Squares算法的方式来将特征点集合自动转换为特征区域的多边形,实现轮廓扫描获取的功能。但是实现的效果嘛,和实际的区域存在着一些较大的差异。 Order by Date Name Attachments HR2MSI mouse urinary bladder S096 - spatial regions […]
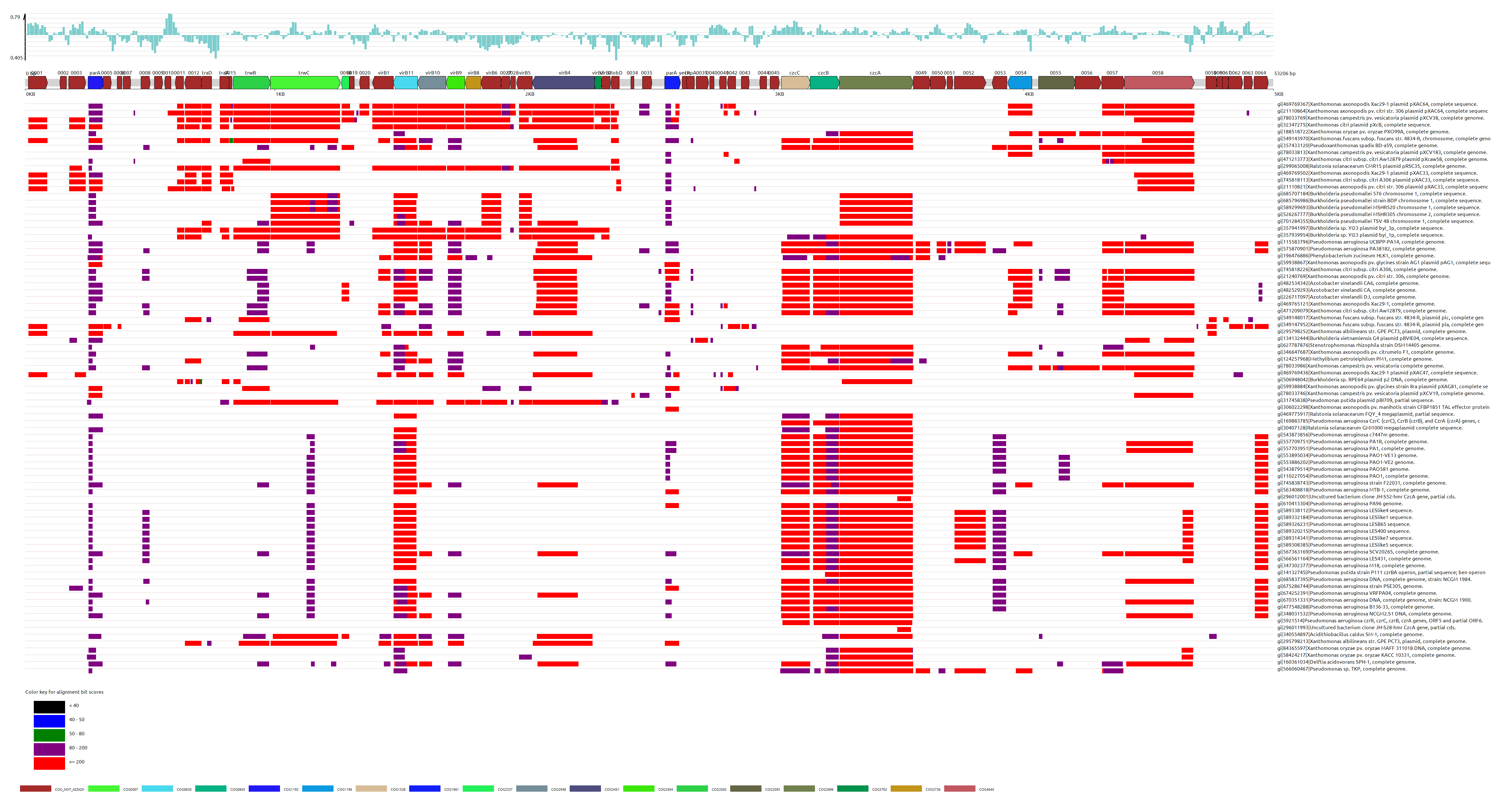
估计阅读时长: 14 分钟在基因组学研究中,将新测序的基因或者针对目标基因组进行基于KEGG代谢通路体系的虚拟细胞建模,都会需要将目标基因组与已知功能基因进行比对注释。KEGG(Kyoto Encyclopedia of Genes and Genomes)数据库通过其KEGG Orthology (KO)系统,为基因功能注释提供了一个强大的平台。KO系统将功能上保守的直系同源基因归为一类,每个KO条目(K编号)代表一个直系同源基因群,这些基因在不同物种中通常执行相似的生物学功能。因此,将新基因的序列与KEGG数据库中的已知基因进行比对,可以推断其可能的KO编号,从而将其功能映射到KEGG通路图或功能层级中。 Order by Date Name Attachments kegg_overview • 313 […]
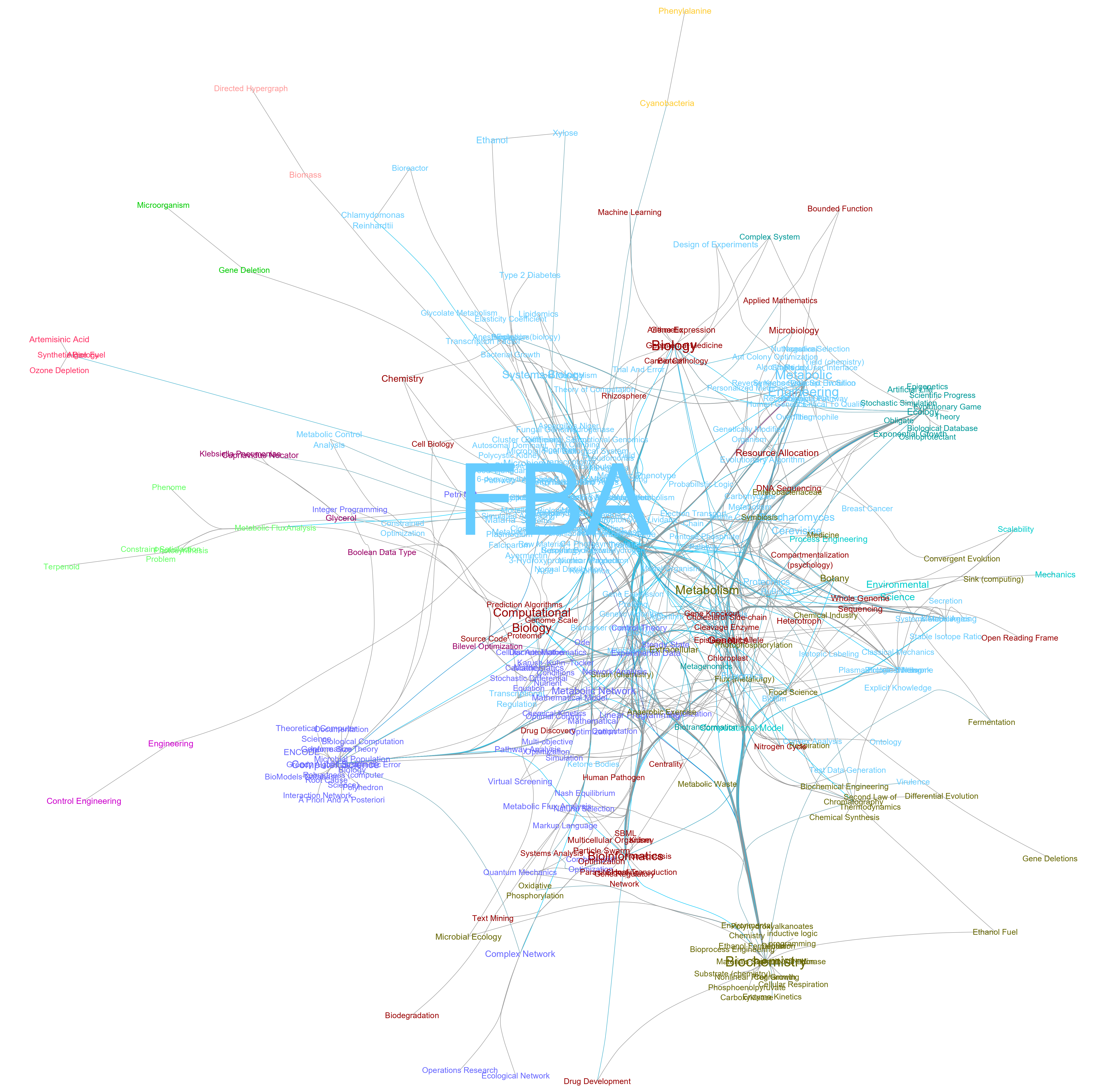
估计阅读时长: 7 分钟https://github.com/rsharp-lang/bing-academic 我们在进行一个新的课题项目开始之前,会需要经历过一个开题立项的报告过程。在这个过程之中,我们需要收集与课题相关的信息,例如相关的知识背景信息,建立出一个与课题相关的知识网络。基于此知识网络进行课题的技术相关概念的梳理。 Order by Date Name Attachments v2-8119594eef2838042df8fb5af0523c0c_720w • 126 kB • 721 click 2021年8月15日v2-96d82f034d4084cf1ed6eb4784f36ed4_r • […]
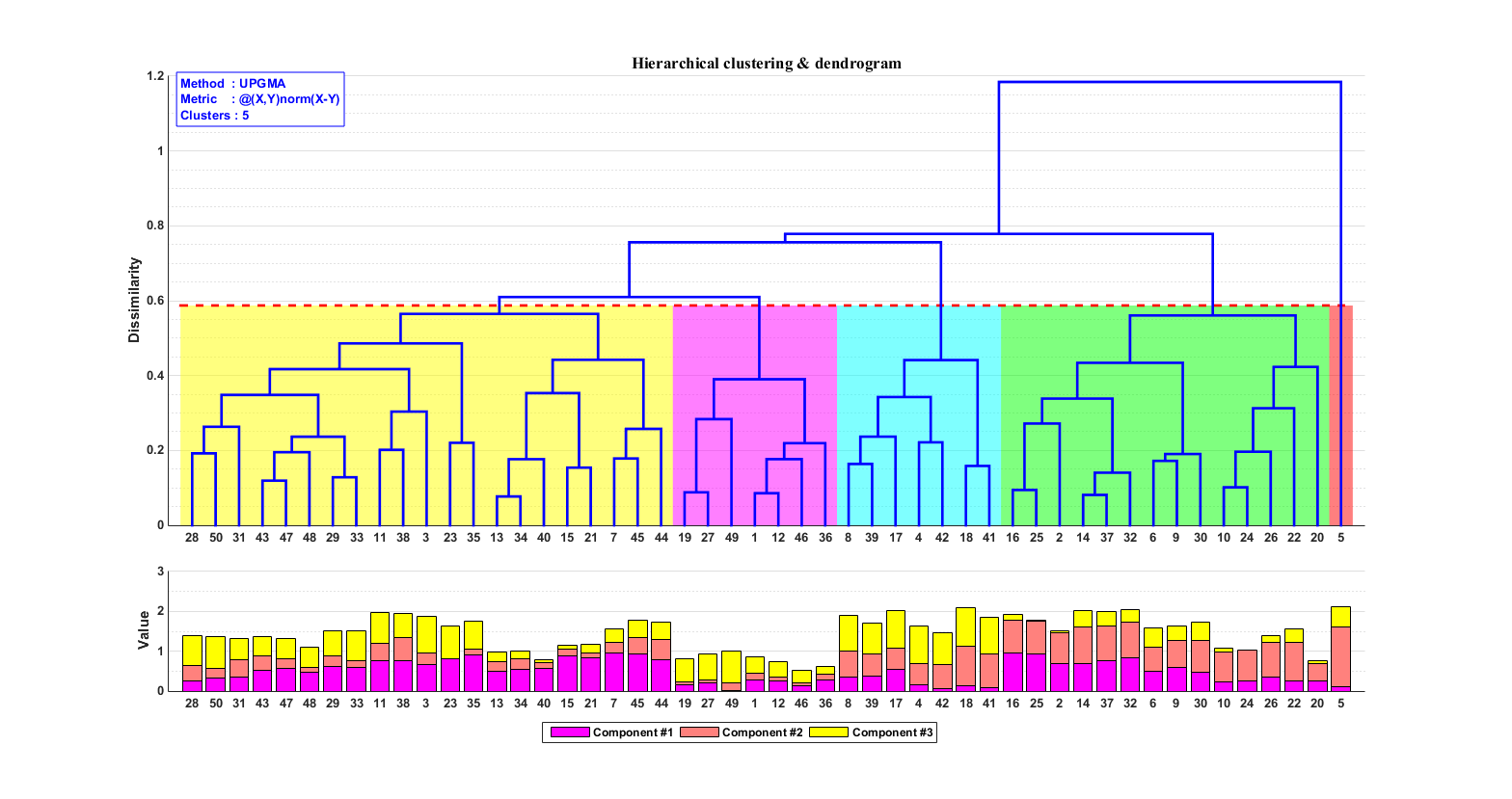
估计阅读时长: 14 分钟https://github.com/xieguigang/sciBASIC 层次聚类通过计算不同类别数据点间的相似度来创建一棵有层次的嵌套聚类树。基于层次聚类分析,我们可以初步可视化我们的一些原始数据: 例如对样本的层次聚类分类,可以让我们了解到样本在分组之间以及分组内的异质性。 对生物序列进行基于相似度的层次聚类分析,我们可以了解到序列之间的相似性程度或者进化关系 Order by Date Name Attachments metabolome • 14 kB • 729 click […]
估计阅读时长: 8 分钟https://github.com/xieguigang/sciBASIC 在进行无监督聚类分析的方法之中,我们在算法代码之中一般会遇到求解与某一个样本数据点最相似的数据点的计算过程。对于这个计算过程,一般而言我们是基于欧几里得距离来完成的。 Order by Date Name Attachments Visual a KDtree Search • 274 kB • 775 […]
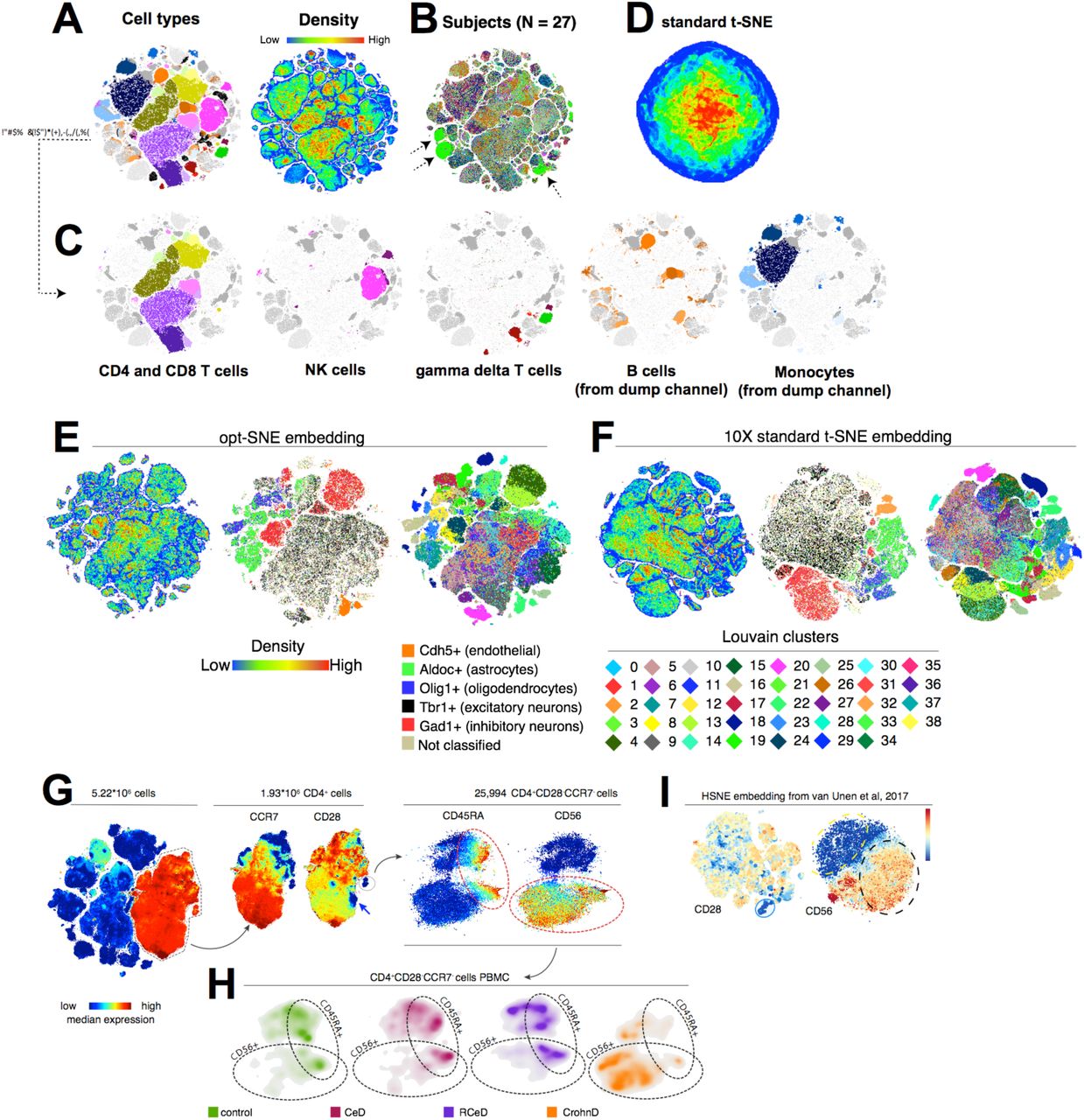
估计阅读时长: 11 分钟PhenoGraph提供了与UMAP类似的算法过程进行单细胞组学数据的细胞分型处理操作。与UMAP方法相比,PhenoGraph并不会产生数据降维效果,仅仅产生数据点Cluster信息。如果需要将数据进行可视化,还需要借助于t-SNE算法将PhenoGraph的分型结果数据投影到一个二维平面上完成。 Order by Date Name Attachments Phenograph-image4 • 200 kB • 711 click 2021年8月9日Automated Optimal Parameters […]
估计阅读时长: 11 分钟https://github.com/xieguigang/sciBASIC Louvain算法是基于模块度的网络节点集群发现算法。该算法在效率和效果上都表现较好,并且能够发现层次性的网络节点集群结构,其优化目标是最大化整个网络集群模块的模块度(Modularity)。 Order by Date Name Attachments graph • 2 MB • 737 click 2021年8月7日Metavirome network […]
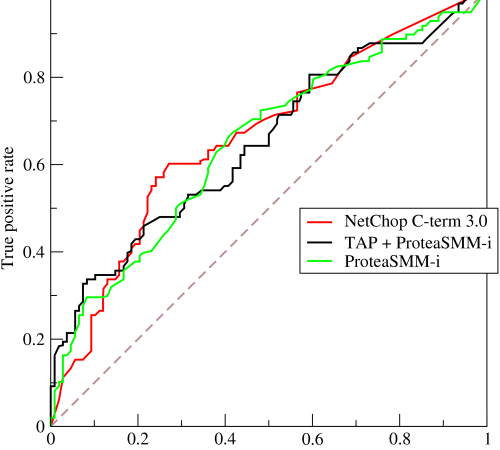
估计阅读时长: 8 分钟https://github.com/rsharp-lang/R-sharp 对于0,1两类分类问题,一些分类器得到的结果往往不是0,1这样的标签。如神经网络得到诸如0.5,0.8这样的分类结果。这时,我们人为取一个阈值,比如0.4,那么小于0.4的归为0类,大于等于0.4的归为1类,可以得到一个分类结果。同样,这个阈值我们可以取0.1或0.2等等。 Order by Date Name Attachments ROC • 221 kB • 786 click 2021年6月28日Roccurves • […]
估计阅读时长: 23 分钟https://github.com/rsharp-lang/R-sharp 降维是将数据由高维约减到低维的过程而用来揭示数据的本质低维结构。它作为克服“维数灾难”的途径在这些相关领域中扮演着重要的角色。在过去的几十年里,有大量的降维方法被不断地提出并被深入研究,其中常用的包括传统的降维算法如PCA和MDS;流形学习算法如UMAP、t-SNE、ISOMAP、LE以及LTSA等。 Order by Date Name Attachments MNIST-LabelledVectorArray-60000x100 • 230 kB • 876 click 2021年6月27日MNIST-LabelledVectorArray-60000x100Euclidean_Distance • […]
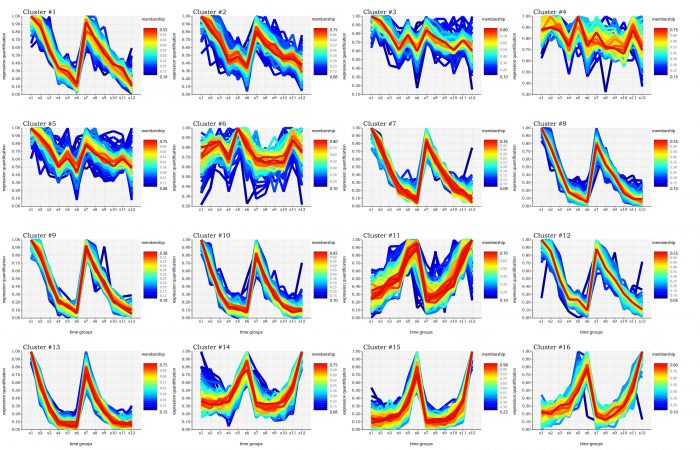
估计阅读时长: 11 分钟https://github.com/SMRUCC/GCModeller 在R语言之中,存在着一个用于进行表达数据的时间序列分析的程序包:TCseq。TCseq的全称为Time course sequencing,即时间序列分析,通过对表达矩阵进行时间上的模糊CMeans聚类,得到表达变化趋势一致的基因列表,进行基因表达的时间趋势分析。 在GCModeller之中,我仿照着TCseq程序包,自己编写了一个时间序列的聚类与可视化分析的R#程序包模块,在这里介绍给大家。 Order by Date Name Attachments Gene expression pattern visualization • 2 […]



[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 我们在基于前面所论述的《通过diamond软件进行blastp搜索》对大规模的基因组数据进行了代谢酶的EC number的注释以及按照文章《基因组功能注释(EC Number)的向量化嵌入》的方法,得到了一个比较大的基因组代谢酶TF-IDF嵌入丰度矩阵后,如果将这里所得到的嵌入结果矩阵中的基因组,基于Family层级的物种分类分组看作为单细胞转录数据中的细胞分群结果,能否基于单细胞数据分析方法来分析和可视化我的基因组功能嵌入的结果矩阵呢? […]
[…] 对于基于ec number来生成层级数据,我们直接使用《酶EC编号结构解析》文章末尾所展示的层级数据生成函数来实现。 […]
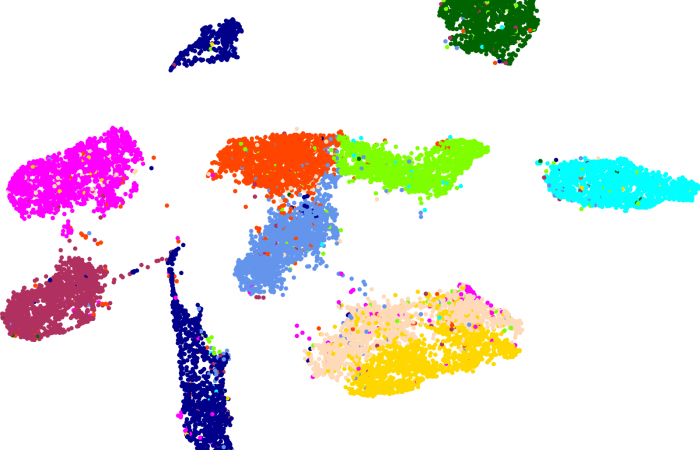
[…] 在前面的一篇《基因组功能注释(EC Number)的向量化嵌入》博客文章中,针对所注释得到的微生物基因组代谢信息,进行基于TF-IDF的向量化嵌入之后。为了可视化向量化嵌入的效果,通过UMAP进行降维,然后基于降维的结果进行散点图可视化。通过散点图可视化可以发现向量化的嵌入结果可以比较好的将不同物种分类来源的微生物基因组区分开来。 […]
😲啊?